降本增效,少了它可不行!
01 背景
随着基因测序技术的不断革新,测序成本普遍下降,测序通量也得到了显著提升。Illumina
NovaSeq 6000 S4 测序芯片能够在 44 小时内产出 > 2,400 Gb的数据,而新近推出的 NovaSeq X Plus 系统更是能够在每次双流动槽运行中产出多达 16,000 Gb 数据量。

图 1. Illumina NovaSeq X 系列及性能参数。
接头是高通量测序文库中不可或缺的关键部分,其中包含的 Index 序列用于区分不同样本,使得单次测序或单个 Flow Cell 通道中允许多个样本混合测序。随着测序通量的增加,单次测序能够容纳的样本量也急剧增加,但是现有市面绝大部分 Index 组合却捉襟见肘,难以满足实际需求。
表 1. 不同应用在满足特定测序深度/数据量下需要的 Index 数量。

* WGS:100 Gb/样本,30x;
** WES:15 Gb/样本,100x;
*** 肿瘤大 Panel:5 Gb/样本,1,000x;
02 Index 设计的重要性
鉴于在文库制备、测序和数据分析等各个环节导致的 Index Hopping、样本交叉污染及背景噪音[1-3]等问题,对 Index 的设计提出了若干要求。一方面,Index 长度的选择需在节省空间以用于样本测序的同时增加容错率[4];另一方面,Index 序列的选择应在很大程度上相互独立,以防止 Index Hopping 引发的样本交叉污染。此外,Index 还应包含一些用于迅速确定检测准确性的代码。最后,Index 设计必须具有灵活性,以满足不同应用之间的细微差异施加的生物学要求:例如:GC 含量、序列冗余、是否存在回文等[4]。
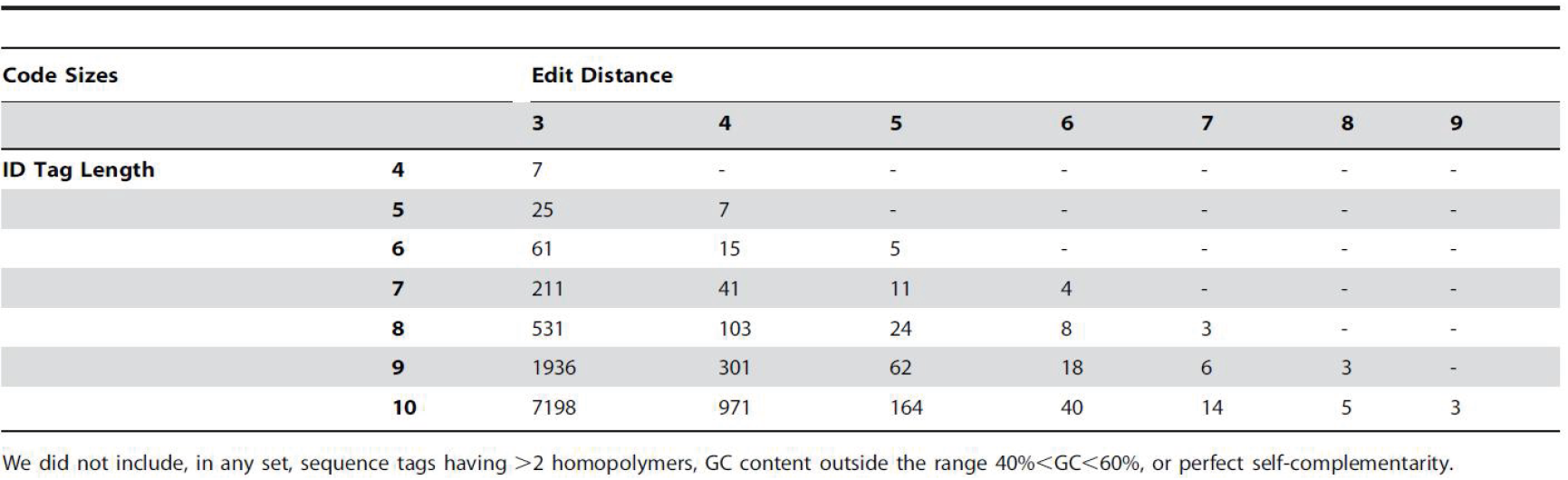
理论上,在满足一定设计要求时,如最小编辑距离 ≥ 3,同源多聚物 ≤ 2、40%< GC < 60% 且不具备自相互补性,不同 Index 序列长度下,允许设计的 Index 种类是有限的。当 Index 序列长度为 6 nt 时最多有 61 种备选;当 Index 序列长度为 8 nt 时最多有531 种备选;而序列长度为 10 nt 时则增加到 7,198 种,可显著提升 Index 的多样性[5]。
表 2. 基于 EDITTAG 工具集设计的序列长度为 4-10 nt 的标签集[5]。
纳昂达推出的 NadPrep®️ Universal Stubby Adapter
(UDI) Module 系列接头模块,其全新设计的双端唯一标签 (10 碱基),可以有效解决测序通量的提高带来的 Index 数量压力以及混合测序过程中可能导致的标签错配等问题。此款接头既兼容目前 Illumina 平台主流的 8 nt Index 测序模式,同时也支持 10 nt Index 测序模式,满足不同应用场景中更多样本混合测序的需求。
03 纳昂达独家专利设计 (专利号:CN 113999893 B)
经优化设计的 NadPrep®️ Universal Stubby Adapter
(UDI) Module 为双端唯一的 10 nt Index (barcode),可支持 Illumina 平台 8 nt 或 10 nt Index 读取模式,且符合 Illumina 平台 8 碱基 4 Index 最小色彩平衡,更易实现多文库混合测序。

图 2. NadPrep®️ Universal Stubby Adapter
(UDI) Module 由通用截短型接头和 UDI Indexing Primer 组成。
表 3. UDI Index Primer 设计原则。

* Index 序列即使在缺失/插入 1 个碱基时依然严格保证 3 个编辑距离。
04 产品表现
4.1 碱基平衡设计优势
NadPrep®️ Universal Stubby Adapter (UDI)
Module 沿用纳昂达双端唯一标签接头设计理念,为 10 nt 4 Index 平衡:≥ 4 个连续 Index 文库等比例混合测序时,Index 中各碱基即可满足平衡,从而有效提高数据的拆分使用。针对 Illumina 平台的多Index 设计方案,例如 Vendor I* 的 UDI Adapter,即使多达 12 个连续 Index,仍无法满足测序要求。
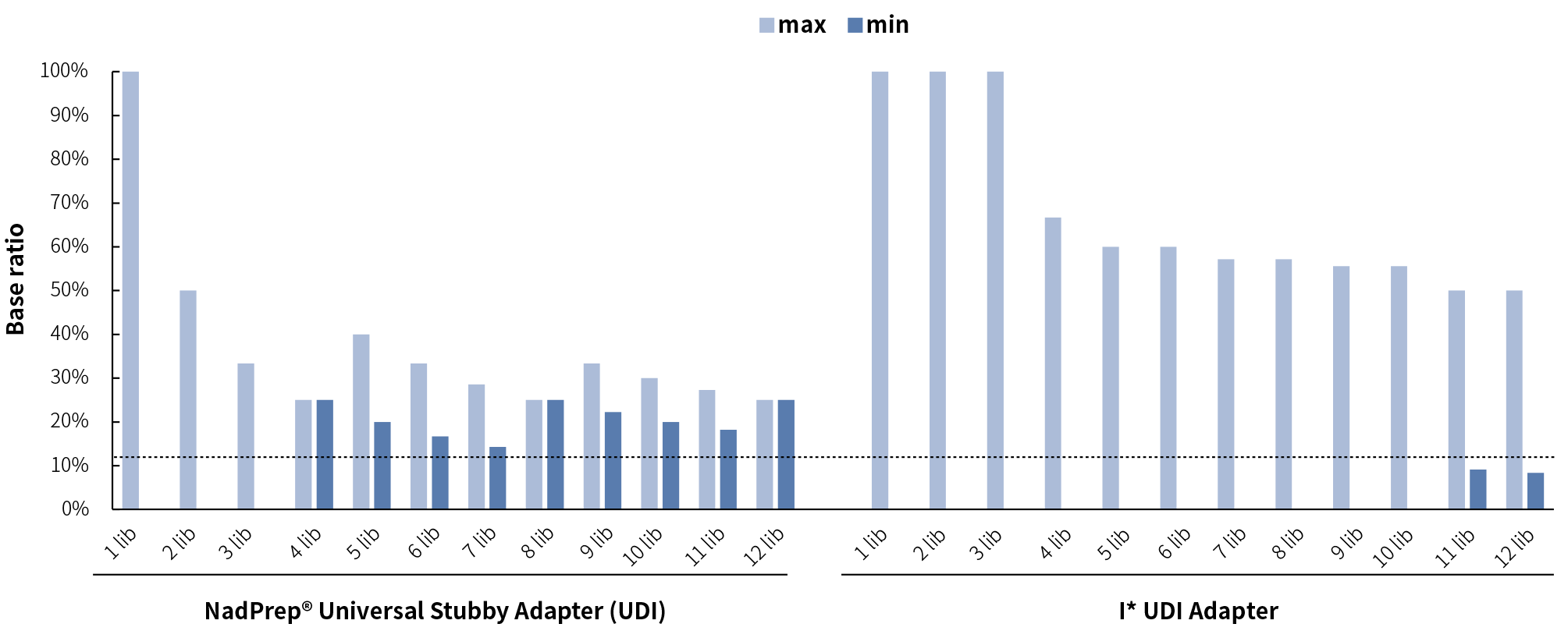
图 3. NadPrep® Universal Stubby Adapter
(UDI) 碱基平衡设计。
4.2 均一的文库和数据产出
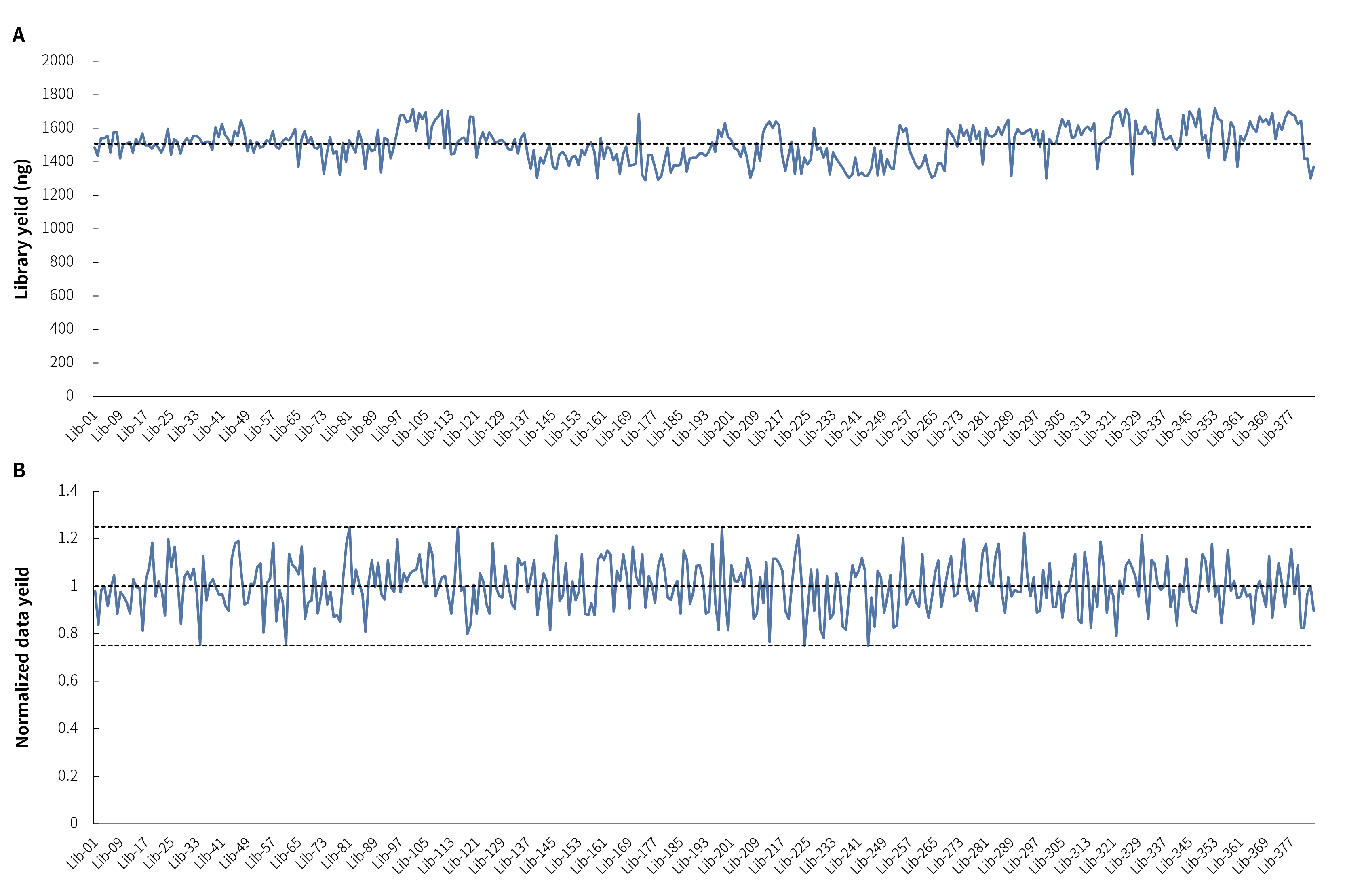
图 4. NadPrep®️ Universal Stubby Adapter
(UDI) 应用示例。
A. 384 个 Universal
Stubby Adapter (UDI) 构建文库的产量。10 ng片段化 gDNA,按 NadPrep® DNA Library Preparation
Module (for Illumina®) 说明书操作,分别使用 Index 1-384,扩增 9 个循环,平均文库产出均 > 1000 ng;B. 384个 Universal Stubby Adapter (UDI) 构建文库在 Illumina 平台的有效拆分率。测序模式:Novaseq 6000,PE150。
4.3 有效提升数据准确度
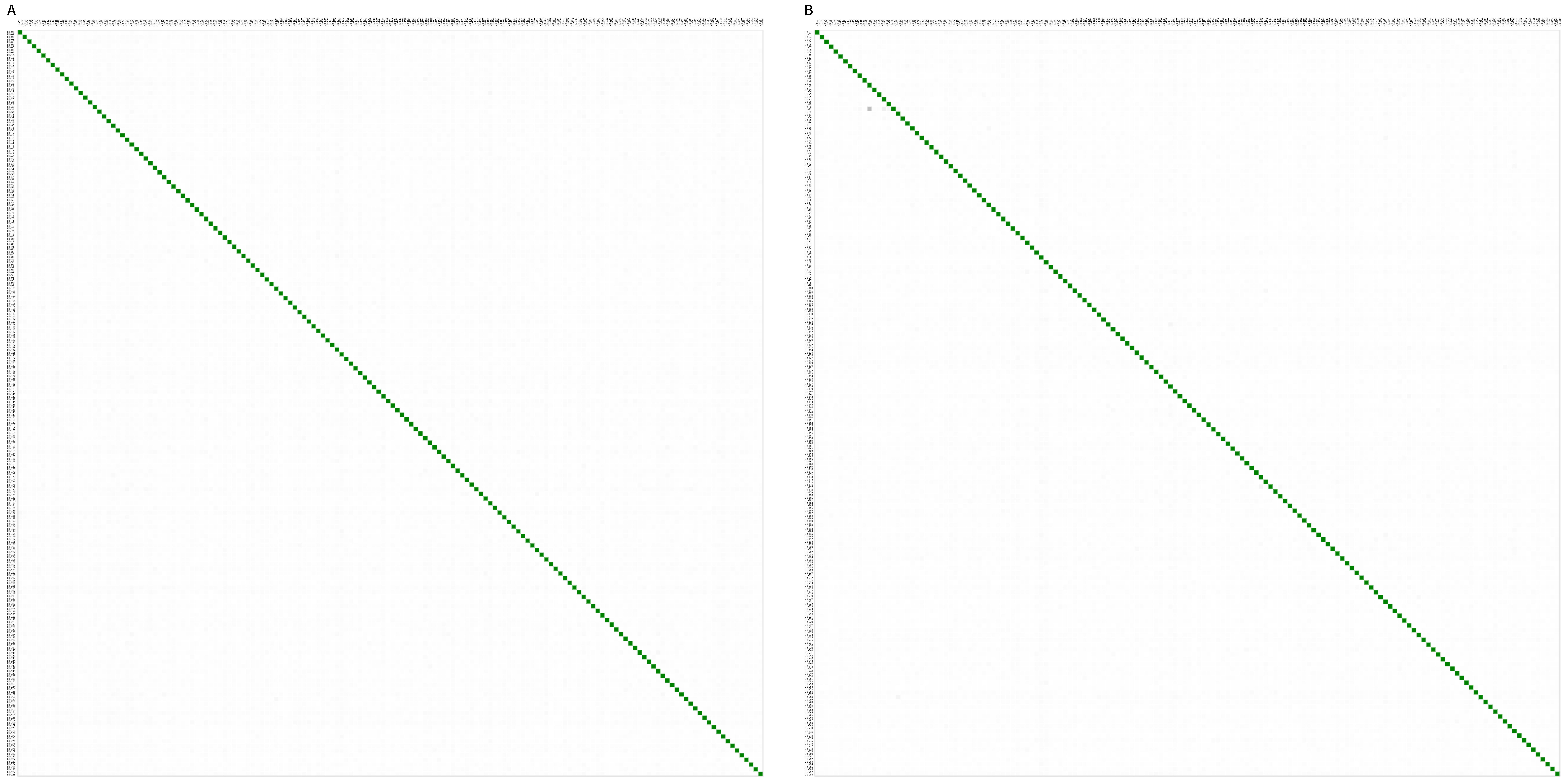
图 5. 数据串扰分析。288 个 Universal Stubby Adapter (UDI) 构建的 WGS 文库,混合后单条 lane 测序。
注:绿色代表 perfectmatch 配对 barcode% > 98.5%。A & B 分别为两个不同批次。
4.4 更高的文库产出
利用 NadPrep® DNA Library Preparation Kit
(for Illumina®) 分别搭配 NadPrep® Universal Stubby Adapter
(UDI) Module 和 NadPrep® UDI Adapter Module (for
Illumina®) 构建文库。在相同的投入量下扩增相同循环数,Universal Stubby
Adapter (UDI) 具有更高的扩增效率,能够高效稳定产出。
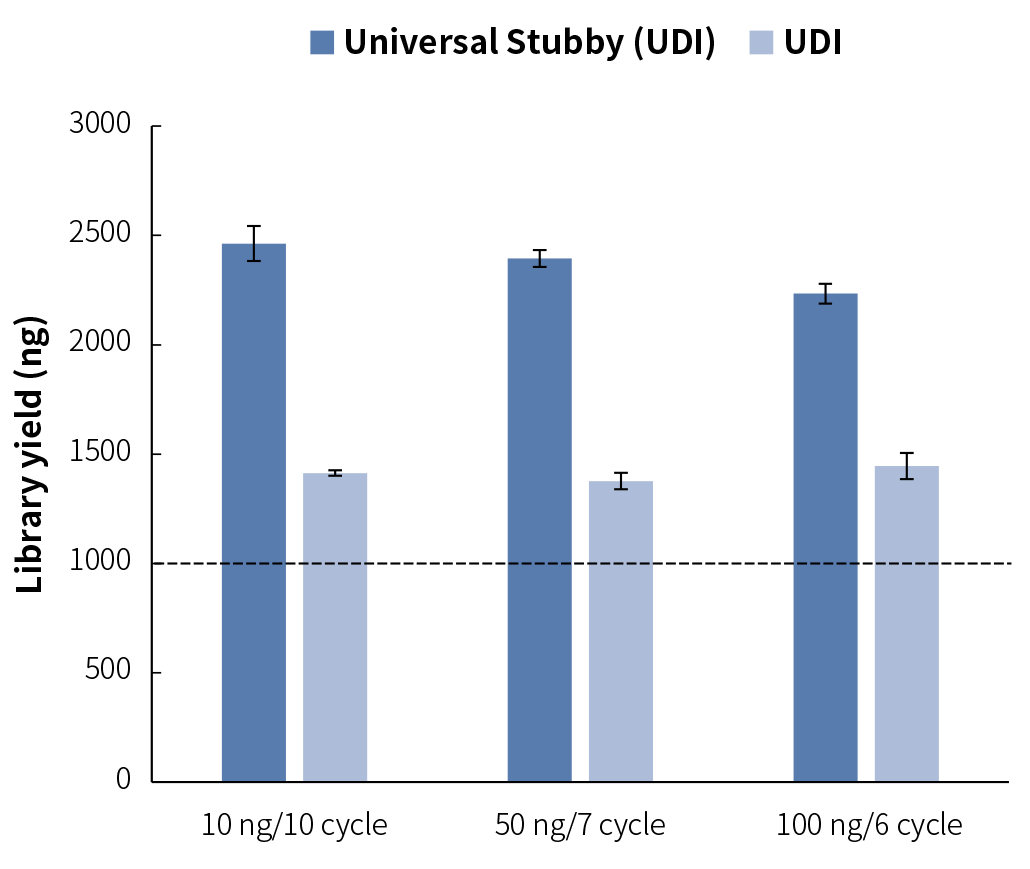
图 6. 搭配不同类型接头的文库产出。NadPrep®
DNA Library Preparation Kit (for Illumina®) 分别搭配 NadPrep® Universal Stubby Adapter (UDI) Module 和 NadPrep® UDI Adapter Module (for Illumina®) 在不同 gDNA 起始量下的文库产出。
注:样本为人类女性基因组 DNA (Promega-Female, G1521)。
4.5 兼容杂交捕获试剂
使用 NadPrep® Universal Stubby Adapter (UDI)
Module 构建的文库可以兼容NadPrep®️ Hybrid Capture Reagents 及 NadPrep®️ NanoBlockers (for Illumina®️)。杂交捕获数据表现与 NadPrep®️ UDI Adapter (for Illumina®) (8 nt) 文库相当。
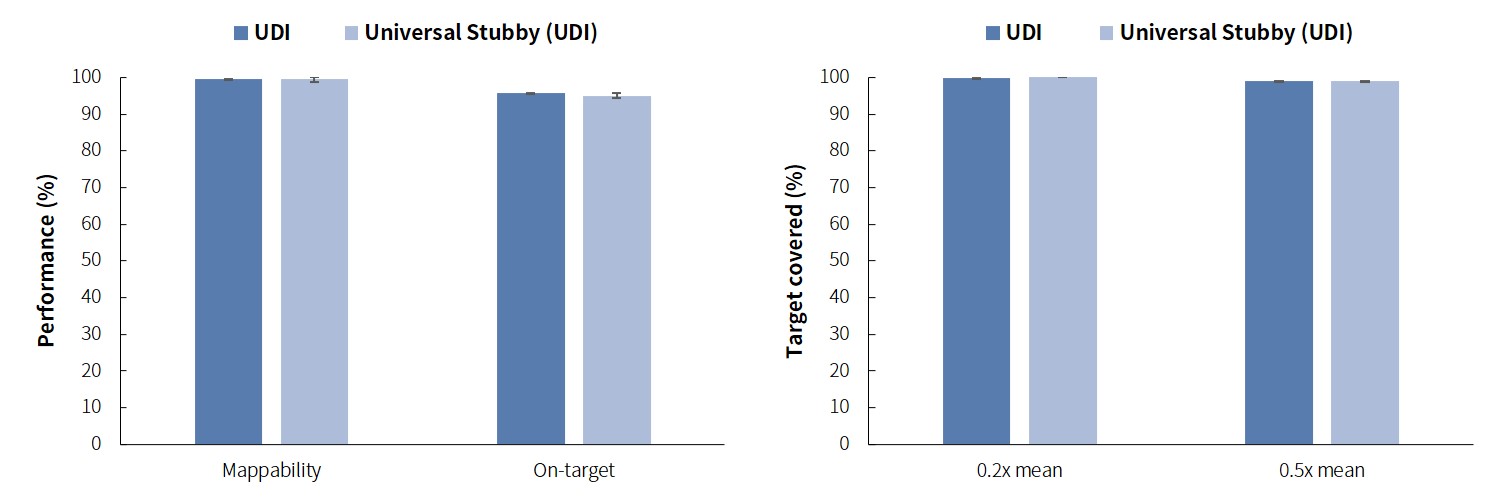
图 7. NadPrep® Universal Stubby Adapter
(UDI) 兼容杂交捕获试剂。人类男性基因组 DNA (Promega-Male, G1471) 利用 NadPrep® DNA Library Preparation Kit (for Illumina®) 分别搭配 NadPrep® UDI Adapter Module (for Illumina®) 和 NadPrep® Universal Stubby Adapter (UDI) Module,以 LungCancer Panel v1.0 (Nanodigmbio, 1001921) 完成杂交捕获。
05 订购信息
参考文献
[1]https://www.illumina.com.cn/content/dam/illuminamarketing/documents/products/whitepapers/index-hopping-white-paper-770-2017-004.pdf?linkId=36607862.
[2] Gilles A, Meglécz E, Pech N, et al.
Accuracy and quality assessment of 454 GS-FLX Titanium pyrosequencing[J]. BMC
genomics, 2011, 12: 1-11.
[3] Nguyen P, Ma J, Pei D, et al.
Identification of errors introduced during high throughput sequencing of the T
cell receptor repertoire[J]. BMC genomics, 2011, 12: 1-13.
[4] Bystrykh L V. Generalized DNA barcode
design based on Hamming codes[J]. PloS one, 2012, 7(5): e36852.
[5] Faircloth B C, Glenn T C. Not all
sequence tags are created equal: designing and validating sequence
identification tags robust to indels[J]. PloS one, 2012,7(8): e42543.








