tNGS 病原检测实例分享
01 背景
宏基因组测序 (mNGS) 作为一种不可知的、公正的、全面的微生物检测和分类学表征方法,已成为一种有吸引力的策略。尽管许多研究和病例报告已证实 mNGS 在改善传染病的诊断、治疗和追踪方面取得了成功,但仍需克服检测灵敏度和成本上的障碍[1-2]。靶向富集测序 (tNGS) 具有富集特定病原体或病原体群以及其他感兴趣基因的特定靶标的优势。tNGS 可在增加样本中病原微生物信息、提高灵敏度的前提下大大减少测序数据量,从而实现性能及成本的双重优化。因此,mNGS 更适用于检测不可预见病原体的场景,而 tNGS 则更适用于分析灵敏度要求更高的场景。相比于 mNGS,tNGS 病原检测有如下技术优点[3-5]:
①不易受人类基因组及背景菌影响
②对 RNA 病毒、胞内菌以及被吞噬病原体检测更灵敏
③可直接针对表型基因检测,如耐药、毒力等相关基因
④可进行病原体真实定量检测
⑤较低的检测成本
近期,纳昂达推出一款全新的基于探针杂交捕获的 tNGS 方案:NEX-t Panel(新品上线 | NEX-t Panel:更精准、更经济的 tNGS 病原检测方案)。该方案靶向数百种病原 (病毒、细菌、真菌、寄生虫) 基因组中的特征序列,此外涵盖 16S/ITS、看家基因、耐药相关基因等区域。与该方案配套的一站式生物信息学分析流程:NEX-Tscan,可用于病原微生物检测分析、16S 和 ITS 全长捕获检测分析。在本文中,我们主要展示其应用于微生物标准品和真实临床样本检测的表现,并与 mNGS 和多重扩增验证比较。
02 材料与方法
2.1 样本来源
微生物标准品:20 菌株交错的混合基因组材料 (ATCC,MSA-1003),其中 13 种微生物基因组在目标靶向物种中设计覆盖,全部 20 种微生物均被 16S 全长探针覆盖。上述标准品经人源背景 gDNA 梯度稀释,得到浓度为 1%、0.1%、0.01% 和 0.001% 等 4 个梯度样品用于测试。
真实样本:21 例真实临床病人样本,均采集自确诊为潜在病原体感染患者。样本类型涵盖组织、尿液、血液、脓液、伤口、脑脊液、肺泡灌洗液、痰液、宫腔分泌物和未知等不同来源。
2.2 实验流程
所有样本经由纳昂达 NadPrep®️ 快速 DNA 酶切文库构建试剂盒 v2 构建预文库,之后使用纳昂达快速杂交试剂和 NEX-t Panel v1.0 捕获,在 Illumina 平台 PE150 测序。为模拟病原检测时常见的 SE50 和 SE100 快速测序策略,我们在原始测序数据基础上通过裁剪读长长度构建 SE100 和 SE50 的数据集,后续进行不同读长数据的对比分析。对于 21 例真实临床样本,我们将同批次提取的 DNA 样本平行委托给第三方分别进行:多重 PCR 测序 (SE60) 和 mNGS (SE100),并与相应的病原体检测报告进行对比分析。
2.3 NEX-tScan 病原数据库
NEX-tScan 基于 NEX-t
Panel 设计策略,通过内部比对打分算法与公开数据库比对,将可捕获的物种序列构建成内部病原体数据库。数据库中的每种病原体均已进行注释分类和人工校对,注释包括细菌、真菌、病毒、寄生虫、支原体衣原体、特殊病原体(结核分枝杆菌复合群)等。此外,数据库也根据公开数据库和文献报道将病原体进行等级划分,包括常见病原、常见环境背景菌、定植菌等,为后续分析和报告提供参考。
03 结果
3.1 微生物标准品
在 1% 和 0.1% 浓度的微生物标准品中,使用 PE150、SE100 和 SE50 三种测序数据模式分析时,靶向捕获探针均可实现 13 种微生物的 100% 检出,且捕获的数据量与各菌种占比相关性 R2 均 >0.7(图 1.
A&B)。同时在这两个浓度下,仅使用靶向 16S 的测序数据分析,也均能检测全部 20 种微生物,相关性 R2 均 >0.7(图 1. C&D)。而在 0.01% 和 0.001% 浓度的微生物标准品中,由于部分菌种的理论拷贝数(图 1. E)接近 0,故未能检测到,检出率有一定降低(图 1. A&C)。对 0.001%~1% 浓度梯度的微生物标准品整体分析结果显示,即使微生物含量低至 3 拷贝,仍能有效检出。
尽管 PE150、SE100和 SE50 三种数据分析模式下,检出结果上无明显差别,但在捕获数据量上存在显著差别。如图 2.所示,由于比对更为精准, PE150 与 SE100 模式检出的读长支持数显著高于 SE50。



图 1. 梯度微生物标准品靶向捕获和 16S 捕获的检出率及相关性分析。A. 专项捕获菌种检出率;B. 专项捕获菌种 read 数量与标准浓度的相关性;C. 16S 菌种检出率;D. 16S 菌种 read 数量与标准浓度的相关性; E. 微生物标准品理论拷贝数。
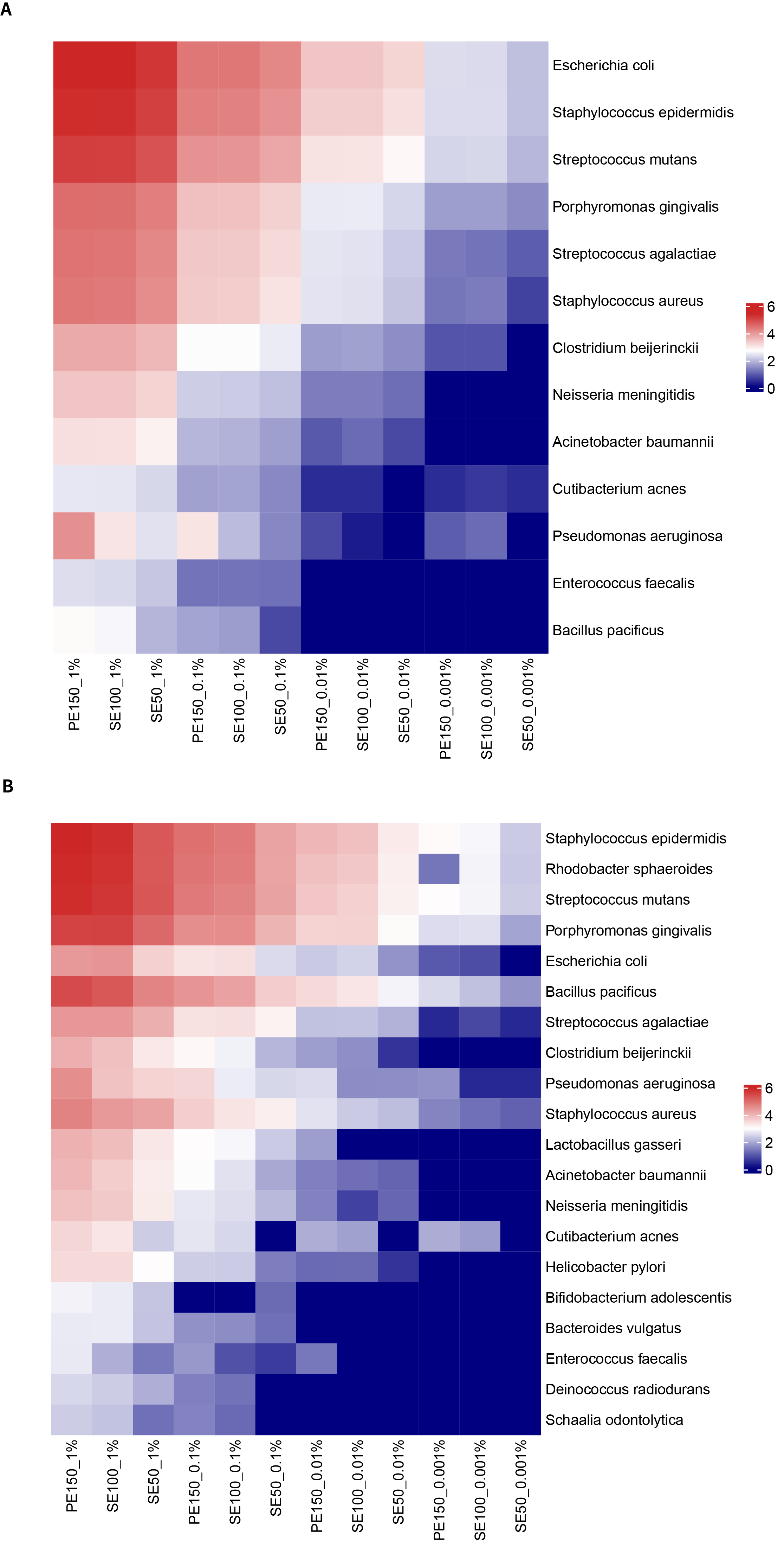
图 2. 梯度微生物标准品在不同测序分析模式下的检出读长支持数(lg,以 10 为底对数)。A. 靶向捕获 13 种细菌数据量热图。B.16S 区域捕获 20 种细菌数据量热图。
3.2 真实样本
不同技术路线的检测结果
我们首先分析比较了 3 种技术路线:纳昂达杂交捕获法 (NEX-t Panel,SE50)、多重 PCR 扩增法和 mNGS 法应用于 21 例真实样本的检测结果。为排除干扰因素,所有分析均基于临床表征不明但组织来源明确的同一样本。同时使用最为严格的判别标准:报告出的致病菌种与“参考结果”完全一致时为正确,少报、多报和错报均定义为错误(表 1.)。
在 21 例真实样本中,多重 PCR 扩增法的准确率为 47.6% (10/21),mNGS 法的准确率为 61.9% (13/21),而杂交捕获法可以达到 71.4% (15/21)。就具体原因而言,多重 PCR 扩增法有 7 例样本的错报属于假阳性错误,4 例样本存在漏报的假阴性错误;mNGS 法则有 4 例假阳性错误,1 例假阴性错误和 3 例完全错误;杂交捕获法则是 5 例假阳性错误和 1 例假阴性错误。
表 1. 不同技术路线病原体检测分析结果。
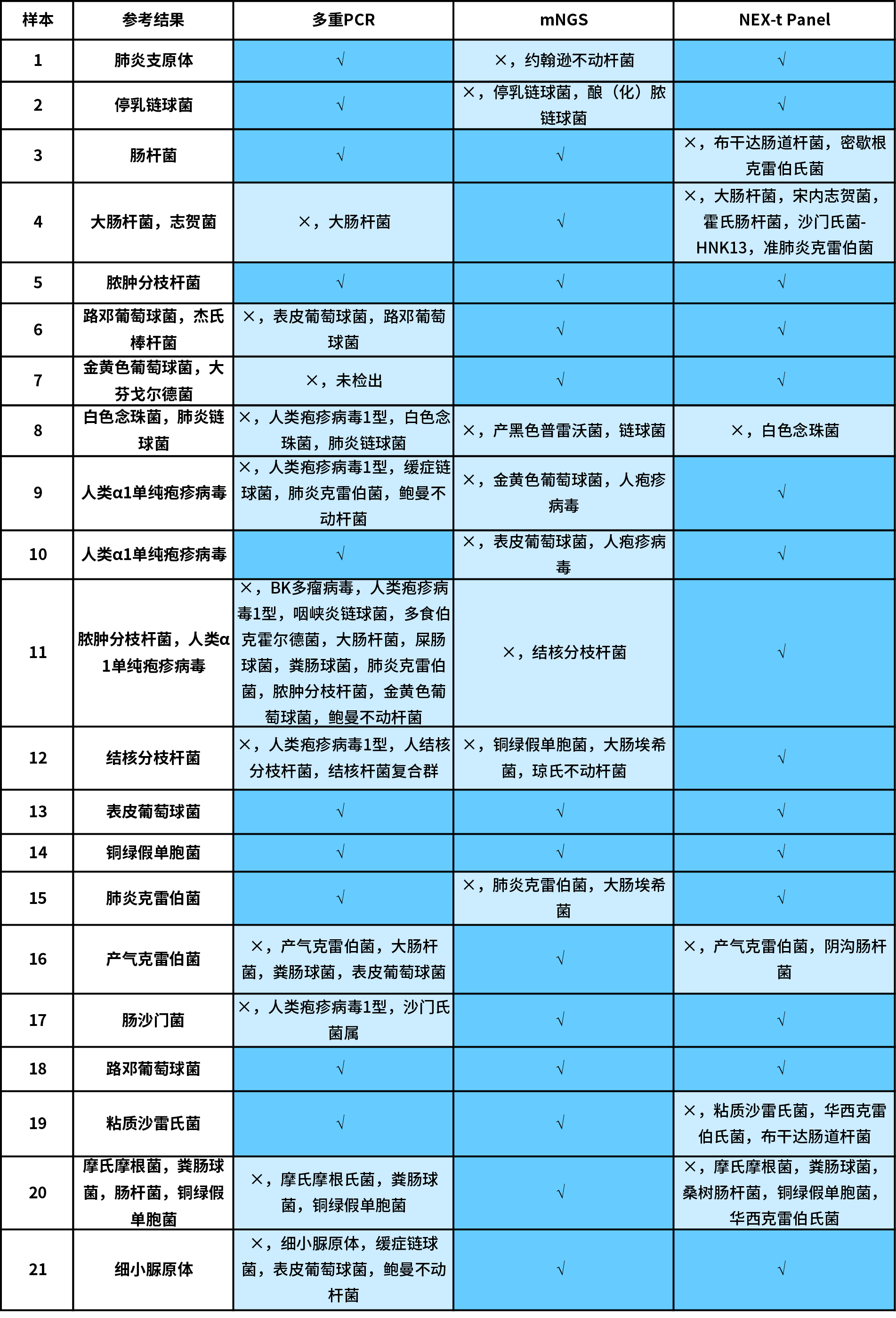
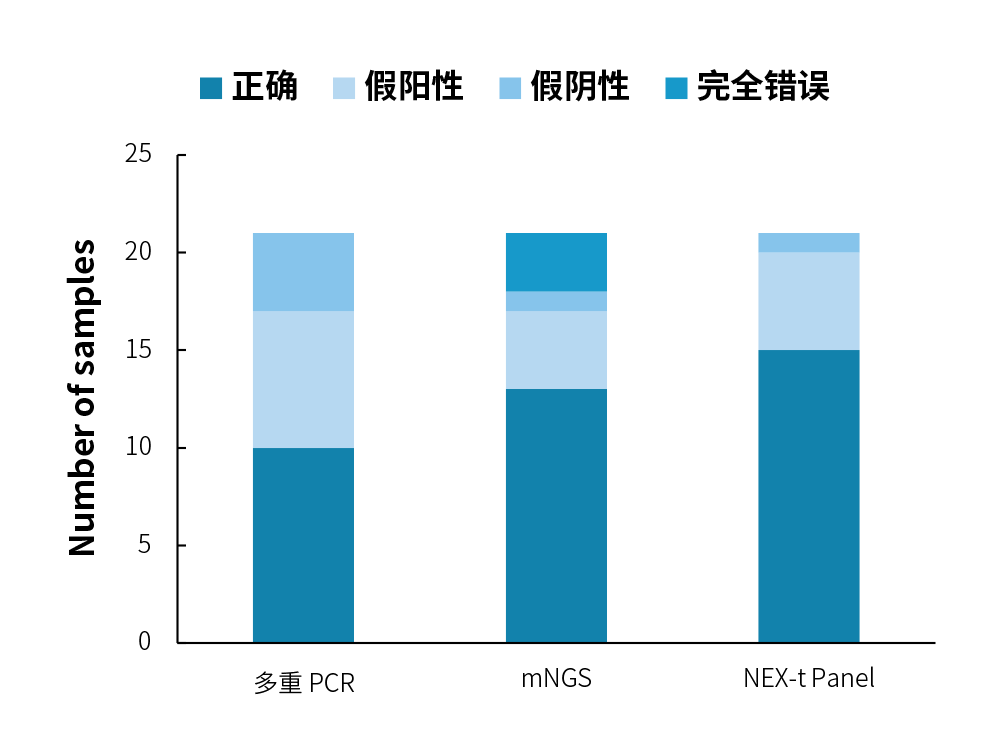
图 3. 不同技术路线病原体检测分析结果汇总。
多重 PCR 扩增法的 7 例假阳性结果中,有近半数(3/7)是由于多报了人类疱疹病毒 I 型,我们推测可能是操作过程中的污染或产品本身缺陷的原因。4 例假阴性结果中的 6 号样本的杰氏棒杆菌及 7 号样本的大芬戈尔德菌可能是由于该款产品未包含相应菌种所致,侧面反映了 PCR 扩增法在检测非常规病原体时的局限性。
mNGS 法中的假阳性结果比较分散,无集中多报某一菌种的现象。而 1 例假阴性和 3 例完全错误则是由于数据本身没有特别优势的菌种或支持读长数量少导致的分析错误,这体现了 mNGS 法数据分析时常遇到的有效数据少分析难度高的问题。
杂交捕获法中的 5 例假阳性结果均集中于克雷伯菌、沙门氏菌、肠杆菌等这几种同源性较高的菌群,这是由于测序读长较短时,探针捕获的区域宽泛性比对所导致。8 号样本这例假阴性结果漏报了肺炎链球菌,是因其占比更接近背景菌。值得注意的是 7 号样本中的大芬戈尔德菌,尽管并未设计专项捕获探针,但是由于 NEX-t Panel v1.0 中包含 16S 探针,使得对其的分析能成功检出。
整体而言,常规病原检测时,多重 PCR 扩增法的引物设计局限性会带来一定的假阴性风险;mNGS 法由于有效数据极难控制及分析难度高,会带来不易判别的假阳性或假阴性风险。杂交捕获法相较前者有更宽泛的菌种检出能力,同时相较后者拥有更加富集的有效数据,因此假阳性和假阴性的检出表现均优于二者。在临床应用时,三种技术路线的初步分析结果,均应由经验丰富的医师结合具体临床表征进一步核准,方能完成最终报告。
进一步提升准确率
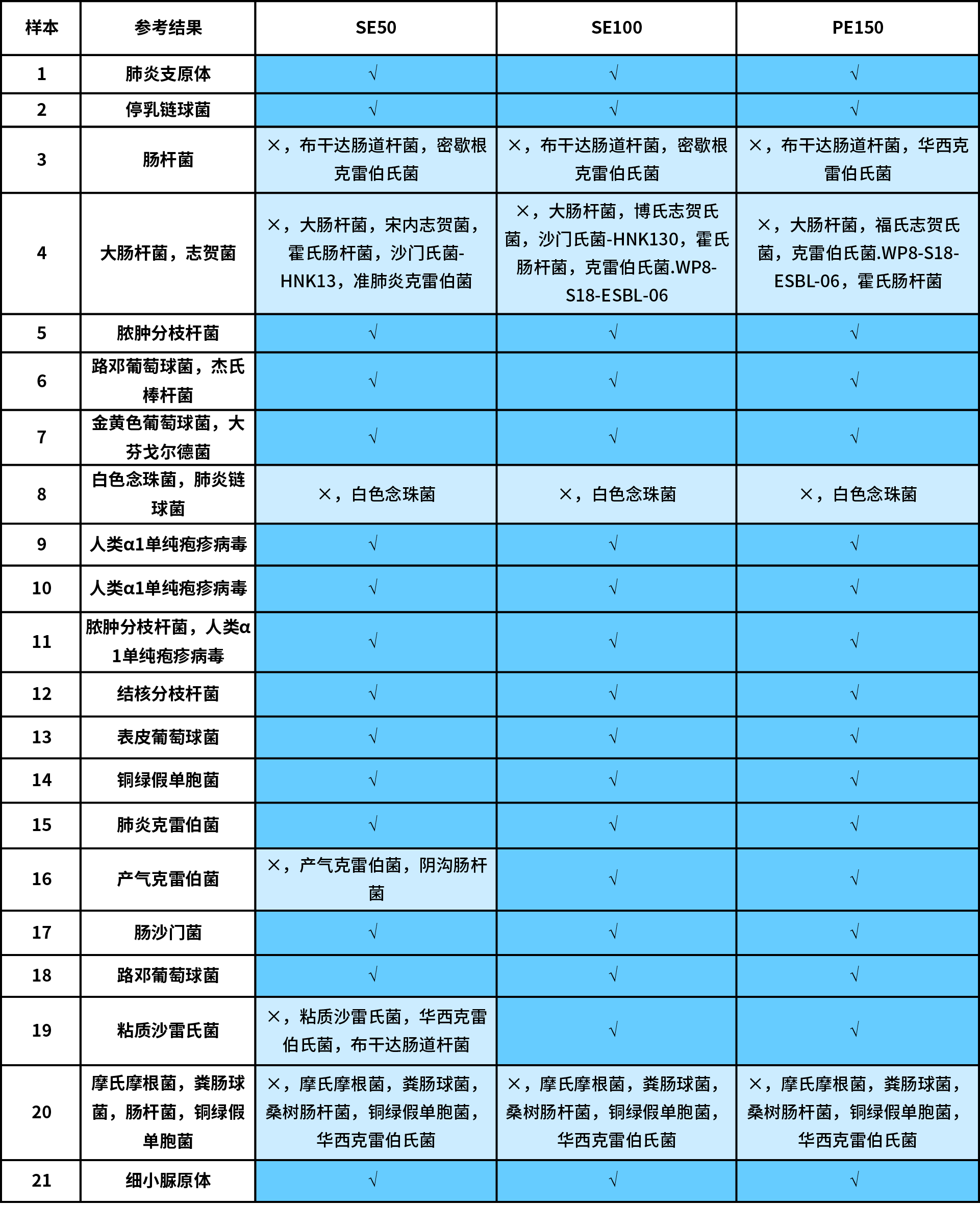
为比较不同测序读长对病原体检测的影响,我们重点评估在 SE50、SE100 和 PE150 三种数据模式下,杂交捕获法的分析结果。整体而言,各样本中在不同测序读长下的检出微生物种类基本一致,说明使用 NEX-t Panel v1.0 捕获后读长长度对病原微生物检出影响有限。
16 号和 19 号样本在 SE50 模式下有明显的假阳性检出,但在 SE100 和 PE150 模式下,检出结果均与参考结果完全一致(表 2.)。这表明,当测序读长较长时,对于同源性较高的菌种的分辨能力更高,将有利于减少假阳性的产生,从而提高结果的正确性。
尽管另外 3 例样本(3 号、4 号和 20 号)的假阳性检出并没有因读长增加而消失,但均是由于肠杆菌科下克雷伯氏菌与其它高度同源菌的划分不明确所致。实际上,像志贺菌和肠杆菌等同源性较高的菌种,往往也需由经验丰富的病原体解读人员从菌种丰度、支持读长数以及临床表型上进行综合判断和解读。
表 2. 不同测序读长分析结果对比。
04 讨论
当临床样本中嵌入了复杂的宿主以及环境复杂背景时,mNGS 可能昂贵得令人望而却步,甚至无法直接通过高通量测序或传统分子技术进行病原微生物的检测。价格低廉、用途广泛且与平台无关的杂交捕获法,在大多数情况下能最大程度上减少微生物基因组测序成本,并能可靠地阐明菌株序列、基因内容和基因组结构。当应用于多菌株鉴定、毒力检测、传播历史追踪或阐明物种进化起源时,杂交捕获法提供给微生物基因组测序效率其他技术无法比拟的[6-7]。
杂交捕获法和多重 PCR 扩增法均可有效富集已知序列,但只有探针杂交捕获可以富集发生了重大重组变异或者 SNP-Indel 突变的序列,比如快速变异的 COVID-19 各种新型毒株。纳昂达推出的 NEX-t Panel v1.0,相对于市场上超大规模杂交捕获 tNGS panel,是一种更经济更便捷的解决方案。尽管 NEX-t Panel v1.0 也存在病原检测范围有限的短板,但是由于杂交探针捕获良好的扩展性,可以根据实际临床检测目的去增加探针,进一步增大检测范围。
05 产品推荐

参考文献
[1] Dahyot S, Lemee L, Pestel-Caron M.
Description et place des techniques bactériologiques dans la prise en charge
des infections pulmonaires [J]. Rev Mal Respir. 2017;34(10):1098-1113.
[2] Ju C R, Lian Q Y, Guan W J, et al.
Metagenomic next-generation sequencing for diagnosing infections in lung
transplant recipients: a retrospective study[J]. Transplant International,
2022, 35: 10265.Gaston, D. C. et al. Journal of clinical microbiology 60,
7(2022).
[3] Gaston D C, Miller H B, Fissel J A, et
al. Evaluation of metagenomic and targeted next-generation sequencing workflows
for detection of respiratory pathogens from bronchoalveolar lavage fluid
specimens[J]. Journal of clinical microbiology, 2022, 60(7): e00526-22.
[4] Campana M G, Hawkins M T R, Henson L H,
et al. Simultaneous identification of host, ectoparasite and pathogen DNA via
in‐solution capture[J]. Molecular Ecology Resources, 2016, 16(5): 1224-1239.
[5] Duggan A T, Perdomo M F,
Piombino-Mascali D, et al. 17th century variola virus reveals the recent
history of smallpox[J]. Current Biology, 2016, 26(24): 3407-3412Forth, J.H. et
al. (2019) A Deep-Sequencing Workflow for the Fast and Efficient Generation of
High-Quality African Swine Fever Virus Whole-Genome Sequences. Viruses.
[6] Forth J H, Forth L F, King J, et al. A
deep-sequencing workflow for the fast and efficient generation of high-quality
African swine fever virus whole-genome sequences[J]. Viruses, 2019, 11(9): 846.
[7] Vezzulli L, Grande C, Tassistro G, et al. Whole-genome enrichment provides deep insights into Vibrio cholerae metagenome from an African river[J]. Microbial ecology, 2017, 73: 734-738.








