精准靶向:长片段 DNA 文库的制备与捕获方案
01 应用背景
近年来,随着精准医学和全基因组研究的不断深入,传统二代测序虽然在高通量和低成本方面具有明显优势,但其短读长 (通常仅 150 ~ 300 bp) 的局限性使其在解析重复序列、复杂结构变异以及高同源区域时显得力不从心。这些区域往往涉及重要的临床相关基因,如肿瘤驱动基因、免疫基因和药物代谢基因,短读长测序难以提供连续完整的信息,导致对大片段插入、缺失以及基因重排等结构变异的检测存在明显不足,同时单倍型解析也只能依赖间接统计推断,难以实现直接相位。
相比之下,三代长片段测序技术凭借其 Kb 级甚至 Mb 级连续读长和不断提升的准确率,为基因组中“暗基因”和单倍型的全面解析提供了全新的解决方案。长读长测序不仅可以跨越复杂的重复区域,还能实现端到端的单倍型组装和结构变异精准定位,从而使得基因组组装、变异检测及疾病关联研究更直观高效。无论是在全基因组 de novo 组装、复杂基因家族解析,还是在对遗传疾病、癌症及免疫相关病变的深入研究中,三代测序均展现出不可替代的优势。
凭借在靶向捕获试剂研发领域的丰富经验,纳昂达针对长读长测序要求,将快速杂交捕获方案成功适配至 Nanopore、CycloneSEQ 和 PacBio 等主流三代测序平台。该方案不仅将捕获文库的插入片段长度拓展至 5 Kb 乃至更长,还保持了优异的中靶率及覆盖均一性,为复杂结构变异、罕见突变以及全长基因组区域的精准检测提供了有力技术保障,同时为临床诊断和个体化治疗策略的制定奠定了坚实基础。此外,该方案沿用二代靶向捕获流程中原有的探针设计,仅替换适用于长片段文库扩增的 PCR 试剂,即可实现从二代到三代长片段测序的无缝技术方案升级。
02 方案流程

03 产品简介
gDNA 片段化
NadPrep® NEM Fragment Module 采用核酸内切酶对 gDNA 进行片段化处理,确保酶切产物末端序列的高完整性,且在酶切过程中不会发生聚合反应。这一过程保留了原有的核酸序列信息和碱基修饰信息,避免了因复制产生的背景噪音。
预文库制备
NadPrep® DNA Library Preparation Kit v2 是基于 A-T 连接原理,针对主流测序平台开发的双链 DNA 文库构建试剂盒。该试剂盒适用于 DNA 的全基因组测序,同时兼容液相杂交靶向捕获测序,在保持高保真度的同时,提升了文库的丰富度。该试剂盒内包含用于 DNA 纯化的磁珠,并适配优化的片段筛选方案;搭配双端唯一样本标签 (Unique Dual Indexes, UDI) 接头 NadPrep® Universal Stubby UDI Adapter Module,可有效改善测序平台的标签跳跃和标签错配问题。
杂交捕获
NadPrep® ES Hybrid Capture Reagents 是针对纳昂达商品化/定制化探针进行靶向捕获快速杂交洗脱步骤优化的试剂盒,以 NadPrep® NanoBlockers 系列和 120 nt NadPanels/NadProbes 系列构成完整的液相杂交捕获体系,支持针对不同大小 Panel 通过调整杂交时间以优化捕获性能,能够在 0.5-4 hr 快速杂交或 16 hr 过夜杂交中灵活选择。
HGBP Panel v1.0 靶向人类血红蛋白编码基因、临近的基因组区域以及调节基因。探针覆盖 160 Kb 基因组区域,用于人类血红蛋白相关的多种变异信息的富集。
DMD Research Panel v1.0 靶向编码抗肌萎缩蛋白 (dystrophin) 的 DMD 基因,覆盖基因组约 2.2 Mb 区域,用于 DMD 完整基因组序列的富集和研究。
NadProbes 定制化探针是纳昂达倾力打造的一站式服务,涵盖从探针设计、生产合成、质检及优化的全流程。通过严格品质控制和优化的 NGS QC 服务,可为客户提供高质量定制化杂交捕获解决方案,覆盖应用类型除常规突变检测外,还包括:甲基化捕获、RNA 捕获、病毒捕获等。
04 方案表现
地中海贫血 gDNA 标准品采用纳昂达长片段 DNA 文库制备和杂交捕获方案构建文库,并经 Nanopore 测序。测序 reads 长度分布主峰约为 6 Kb (图 1.);数据产出均衡,平均中靶率达 50% 以上 (表 1.),与预期结果保持一致。
4.1 测序读长分布
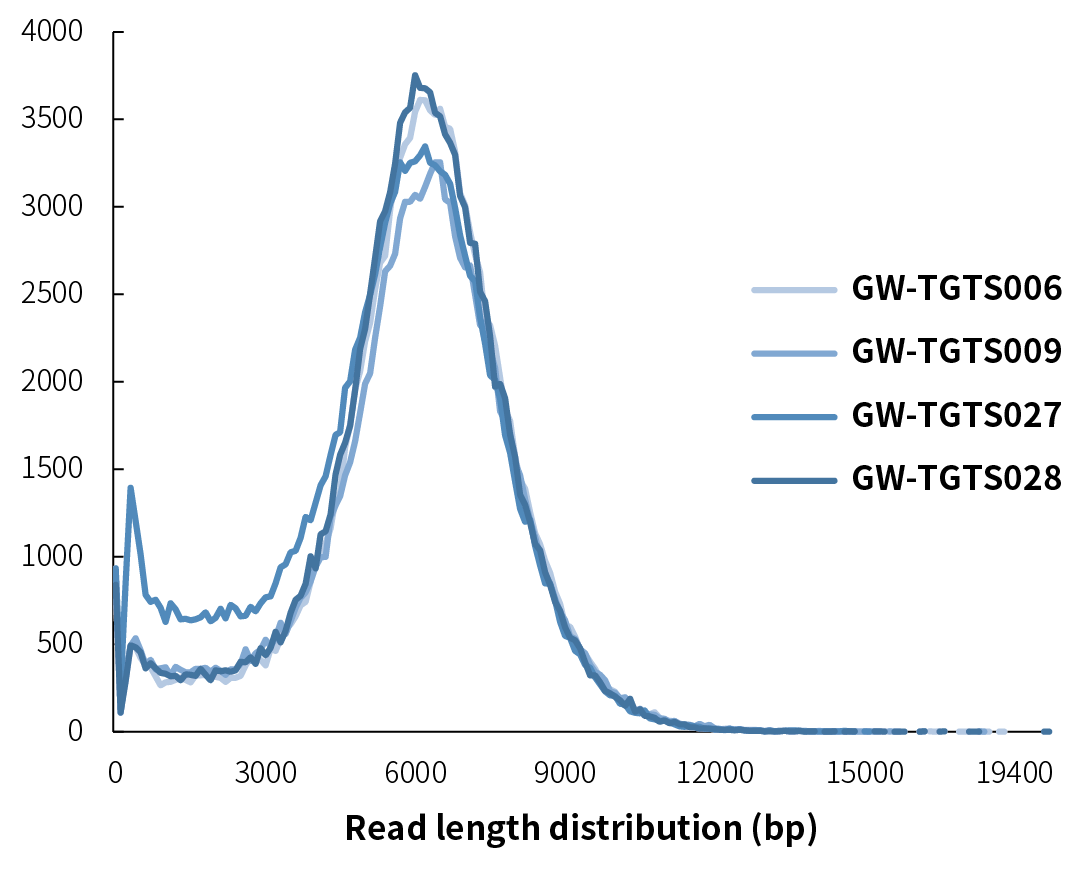
图 1. 测序读长主峰分布于 6 Kb 左右。100 ng gDNA 样本参考长片段 DNA 文库制备和杂交捕获使用说明书构建长片段 DNA 文库,200 ng/预文库投入,以 HGBP Panel v1.0 完成杂交捕获,在 Oxford Nanopore 平台完成长读长测序。
注:样本来源于地中海贫血 gDNA 标准品 (GeneWell),分别为 GW-TGTS006、GW-TGTS009、GW-TGTS027 和 GW-TGTS028。
表 1. HGBP Panel v1.0 对地中海贫血 gDNA 标准品的靶向捕获效率分析汇总。

4.3 目标区域覆盖
应用示例 1:地中海贫血检测 —— HGBP Panel v1.0
许多地中海贫血相关的结构变异来源于同源基因间的重组,而这些基因序列高度相似使得短读长测序生成的 Reads 在基因组比对时无法唯一定位,从而影响结构变异分析的准确性。相比之下,长读长测序能提供更连续的序列,跨越同源区域和复杂重组事件,准确呈现结构变异的真实情况[1-3]。
借助长读长测序平台,HGBP Panel v1.0 对目标区域的覆盖度得到了显著提升 (图 2.)。传统短读长测序仅局限于捕获探针覆盖及侧翼临近区域,且在部分特殊区域存在覆盖不足的问题;而长读长测序则实现了整个基因座的完整均一覆盖,不仅提高了结构变异检测的敏感性,也为血红蛋白基因检测提供了更可靠的技术支持。

图 2. 地中海贫血 gDNA 标准品以 HGBP Panel v1.0 靶向捕获后经长读长测序的目标区域覆盖度优于短读长测序。A & C. 地中海贫血 α 缺失 gDNA 标准品 (GW-TGTS006 和 GW-TGTS009);B & D. 地中海贫血 β 缺失 gDNA 标准品 (GW-TGTS027 和 GW-TGTS028)。
注:A & B 测序平台为:Oxford Nanopore;C & D 测序平台为:CycloneSEQ-WT02。
应用示例 2:DMD 相关遗传疾病检测 —— DMD Research Panel v1.0
DMD 基因位于 Xp21.2,编码抗肌萎缩蛋白,全长超过 2.2 Mb,约占人类基因组的 0.1% 或 X 染色体的 1.5%[4]。DMD 基因序列中 99% 为内含子区域,86 个外显子仅占约 1%。超长序列长度和复杂的外显子结构,使得 DMD 基因的突变发生率远高于其他单基因病的致病基因[5]。DMD 基因完全或部分失功能变异会导致多种抗肌萎缩蛋白同源异构体的异常表达[6],诱发 Duchenne 型肌营养不良 (Duchenne muscular dystrophy,DMD)、Becker 型肌营养不良 (Becker muscular dystrophy,BMD)、X 连锁扩张型心肌病 (X linked dilated cardiomyopathy,XLDCM) 等严重遗传疾病。
在 DMD 基因的内含子序列中存在着丰富的散在重复元件和脆性位点,使其极易出现外显子缺失/重复以及复杂结构变异[7-12]。然而,由于内含子区域存在难以设计探针的危险区域,短读长测序往往出现覆盖缺口的问题,极端情况下甚至可能导致关键断点无法被捕获。借助长读长测序平台,DMD Research Panel v1.0 能跨越高重复和复杂结构区域,实现 100% 的均一覆盖 (图 3.)。对于 DMD 等高度复杂、突变多样的基因,长读长测序不仅消除了覆盖缺口的问题,还能精准解析大规模结构变异和外显子重排,为 DMD 相关遗传疾病的全面检测、精准诊断和个体化治疗提供强有力的技术支撑。
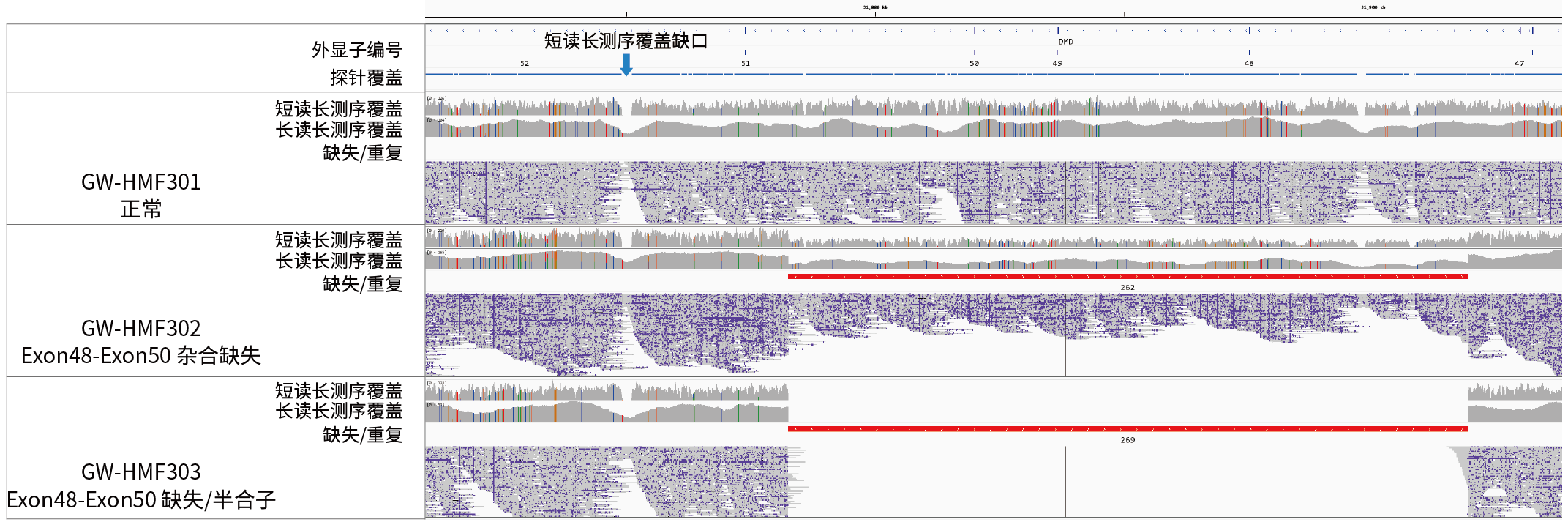
图 3. 杜氏肌营养不良基因检测 gDNA 参考品以 DMD Research Panel v1.0 靶向捕获后经长读长测序实现了 100% 全长覆盖。
注:样本来源于杜氏肌营养不良基因检测 gDNA 参考品 (GeneWell, GW-HMF301-3),分别为家系中 Male (正常)、Female (Exon48-Exon50 杂合缺失) 和 Male (Exon48-Exon50 缺失/半合子)。短读长测序为:NovaSeq 6000,PE150;长读长测序为:Oxford Nanopore。
参考文献
[1] Borg J, Georgitsi M, Aleporou-Marinou V, et al. Genetic recombination as a major cause of mutagenesis in the human globin gene clusters[J]. Clinical biochemistry, 2009, 42(18): 1839-1850.
[2] Li W, Ye Y. Application of third-generation sequencing technology in the genetic testing of thalassemia[J]. Molecular Cytogenetics, 2024, 17(1): 32.
[3] Huang W, Qu S, Qin Q, et al. Nanopore third-generation sequencing for comprehensive analysis of hemoglobinopathy variants[J]. Clinical chemistry, 2023, 69(9): 1062-1071.
[4] Muntoni F, Torelli S, Ferlini A. Dystrophin and mutations: one gene, several proteins, multiple phenotypes[J]. The Lancet Neurology, 2003, 2(12): 731-740.
[5] Tuffery-Giraud S, Miro J, Koenig M, et al. Normal and altered pre-mRNA processing in the DMD gene[J]. Human genetics, 2017, 136: 1155-1172.
[6] Kunkel L M, co-authors. Analysis of deletions in DNA from patients with Becker and Duchenne muscular dystrophy[J]. Nature, 1986, 322(6074): 73-77.
[7] McNaughton J C, Hughes G, Jones W A, et al. The evolution of an intron: analysis of a long, deletion-prone intron in the human dystrophin gene[J]. Genomics, 1997, 40(2): 294-304.
[8] Pozzoli U, Elgar G, Cagliani R, et al. Comparative analysis of vertebrate dystrophin loci indicate intron gigantism as a common feature[J]. Genome research, 2003, 13(5): 764-772.
[9] Oshima J, Magner D B, Lee J A, et al. Regional genomic instability predisposes to complex dystrophin gene rearrangements[J]. Human genetics, 2009, 126: 411-423.
[10] Gonçalves A, Oliveira J, Coelho T, et al. Exonization of an intronic LINE-1 element causing becker muscular dystrophy as a novel mutational mechanism in dystrophin gene[J]. Genes, 2017, 8(10): 253.
[11] Ling C, Dai Y, Fang L, et al. Exonic rearrangements in DMD in Chinese Han individuals affected with Duchenne and Becker muscular dystrophies[J]. Human Mutation, 2020, 41(3): 668-677.
[12] Casper A M, Nghiem P, Arlt M F, et al. ATR regulates fragile site stability[J]. Cell, 2002, 111(6): 779-789.








