完整标题:Demo 数据中测序模式为 PE150,若采用 SE50/75 测序模式是否会对结果判定产生影响?是否需要进一步增加测序数据量?
在病原检测过程中,SE 模式更节省时间,因此比 PE 模式更常用。不管选用何种测序模式,只要检测到足量的 reads,均可用于病原分析,不太会影响最终的结果判定。若选用 SE50/75 测序模式,不需要增加测序数据量。目前的 Demo 数据中实际测序模式为 PE150,可模拟为 SE50 或 SE100 的数据进行分析,对最终的结果判定影响不会太大。
完整标题:NEX-t Panel v1.0 在探针设计层面具有哪些优势,如何平衡捕获性能与经济成本?

- 一般来说,对病原微生物基因组进行完整的探针覆盖是无法实现的。一方面是由于在考虑参考基因组以外的序列多态性时,一种细菌所需的探针即可达数十万条;另一方面是由于当病原序列与宿主序列存在一定相似性时,为防止脱靶,需要舍弃这些区域。因此,NEX-t Panel v1.0 在探针设计时侧重于以最少的探针实现最多的病原分析功能,panel 规模相比于常见的杂交捕获 tNGS 方案大为精简。
- 探针条数可近似等效于多重扩增子的扩增子个数,但由于探针具有更高的容错性,同时还能捕获到探针侧翼的序列;因此,一定数量的探针可获得比相似扩增子个数的多重扩增更丰富的信息。
- 与多重扩增子相比,杂交捕获 Panel 的扩展更为简单,只需增加探针。因此,NEX-t Panel v1.0 可以方便地进行定制和组合升级。

完整标题:NEX-t Panel v1.0 如何缩短 TAT,提升杂交捕获操作便捷性?
- NEX-t Panel v1.0 推荐与 NadPrep®️ ES Hybrid Capture Reagents 快杂试剂搭配,可有效缩短杂交时间,简化实验步骤;
- 杂交捕获测序比多重扩增子测序更为稳定,失败率更低。
完整标题:NEX-t Panel v1.0 捕获中靶率表现如何?是否需要进行两轮杂交来提高中靶率?
- 病原捕获的中靶率受到病原含量的影响较大。如下图所示,在相同的宿主背景下,随着微生物参考品的梯度稀释,其数据量呈等比例下降的趋势,而中靶率也随之降低。
- 尽管多轮捕获能够显著地提高中靶率,但这种策略同时伴随着信息丢失 (dropout) 的风险。尤其在病原含量较低的情况下,中靶率低,同时也对 dropout 更为敏感。因此,在考虑多轮捕获时需要谨慎权衡利弊。此外,从另一个角度来看,当样本中的病原基因组拷贝数为个位数时,提高中靶率实际上只是将这几个拷贝反复测序,并未产生更多的有用信息。
 注:以人类基因组
DNA 标准品
(Promega,G1471)
按照不同比例对 20 菌株交错的混合基因组材料
(ATCC,MSA-1003) 进行稀释以获得 0.001%-1% MSA-1003 的模拟菌群样本。50 ng Input 以 NadPrep® 快速
DNA 酶切文库构建试剂盒
v2 建库,利用
NEX-t Panel v1.0 搭配 NadPrep®️ ES Hybrid Capture Reagents 完成杂交捕获 (杂交
2 h)。测序模式:Novaseq6000,
PE150。
注:以人类基因组
DNA 标准品
(Promega,G1471)
按照不同比例对 20 菌株交错的混合基因组材料
(ATCC,MSA-1003) 进行稀释以获得 0.001%-1% MSA-1003 的模拟菌群样本。50 ng Input 以 NadPrep® 快速
DNA 酶切文库构建试剂盒
v2 建库,利用
NEX-t Panel v1.0 搭配 NadPrep®️ ES Hybrid Capture Reagents 完成杂交捕获 (杂交
2 h)。测序模式:Novaseq6000,
PE150。
测序数据利用 BWA 比对到由 hg19 人类基因组和 20 种微生物基因组参考序列组成的参考基因组,统计 reads 分布。
完整标题:NEX-t Panel 检测灵敏度表现如何?
- 如下图所示,对于不同微生物含量的样本,检出下限均在 3 拷贝附近。实际使用时,灵敏度主要由建库投入量决定。需要注意的是,文库制备存在转化率的制约因素,不是所有拷贝都能成功转化。与之类似,当拷贝数为个位数时,多重扩增子的扩增成功率也会降低。
- 此外,拷贝数小于 1 并不意味着无法被检出,这是因为 NEX-t Panel v1.0 在一个物种存在多个探针捕获区域时,其拷贝数可以累加,从而提高检测灵敏度。

完整标题:样本中的病原丰度各异,NEX-t Panel v1.0 使用时是否需要单独杂交捕获来保证获得相同的数据量?
由于不同样本中的病原含量差异较大,如果希望每个样本都获得相似的数据量,则无法混合杂交,只能单独杂交。不过如下图所示,当样本中微生物含量存在数量级的差异时,虽然数据量差异较大,但是对于低丰度的样本,其本身所需的数据量也相对较低。同时,不同样本之间由于使用相同的实验参数,具有相似的 PCR 重复率。如果将它们分别杂交捕获,并设置相同的测序量,则低丰度样本需要增加 PCR 循环数,相同的信息量只会以重复的形式组成提升的测序数据量,这是因为 Panel 捕获后测序更容易达到饱和。因此,对于具有相似来源的样本,我们建议是可以进行混合杂交的。

完整标题:NEX-t Panel v1.0 是否支持 RNA/DNA 共捕获?
- 支持。RNA 病毒和 DNA 病毒可以采用共建库的方式制备预文库,然后再进行捕获;也可以分别制备 RNA 文库和 DNA 文库,混合后杂交。一般来说 RNA 文库不需要进行宿主核糖体 RNA 的去除。
- 下图示例展示了含有流感病毒 (H10N3) 的 RNA 文库与 DNA 文库共杂交的捕获情况。共计 15 个文库中包含了 11 个 DNA 文库和 4 个 RNA 文库。示例文库数据量为 100 Mb (15 个文库总数据量为 9.3 Gb),其中宿主序列占比为 58.3%。

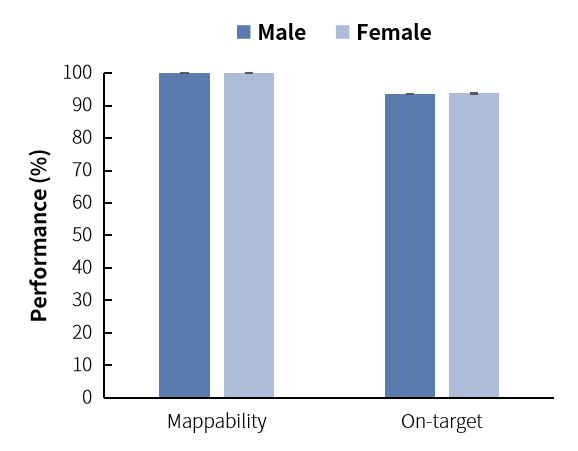
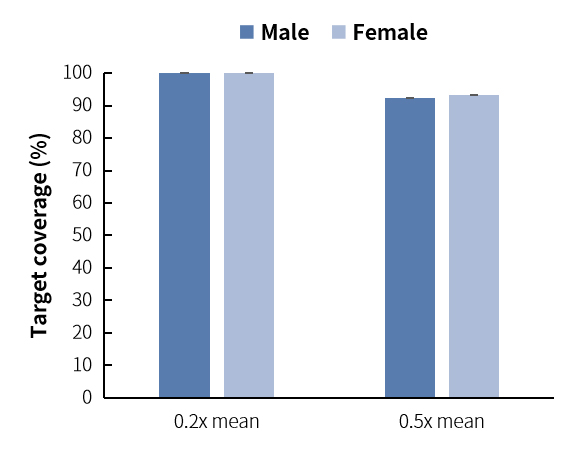
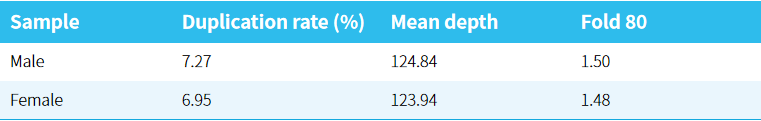
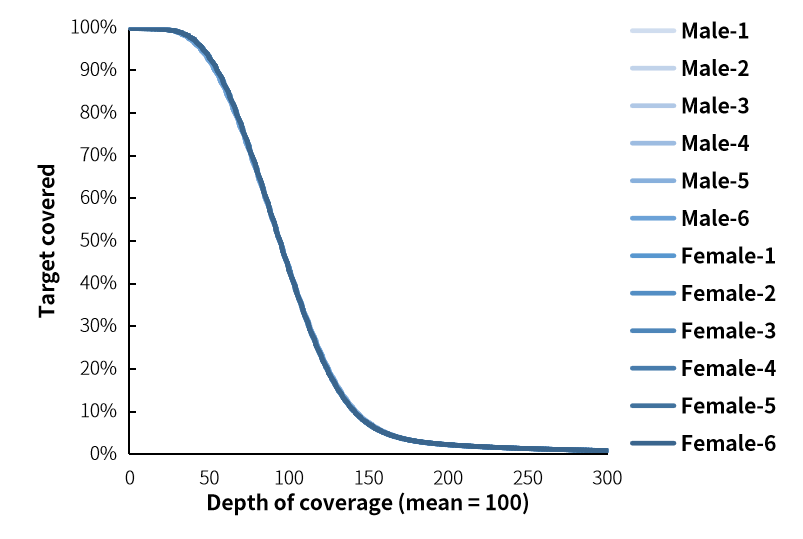
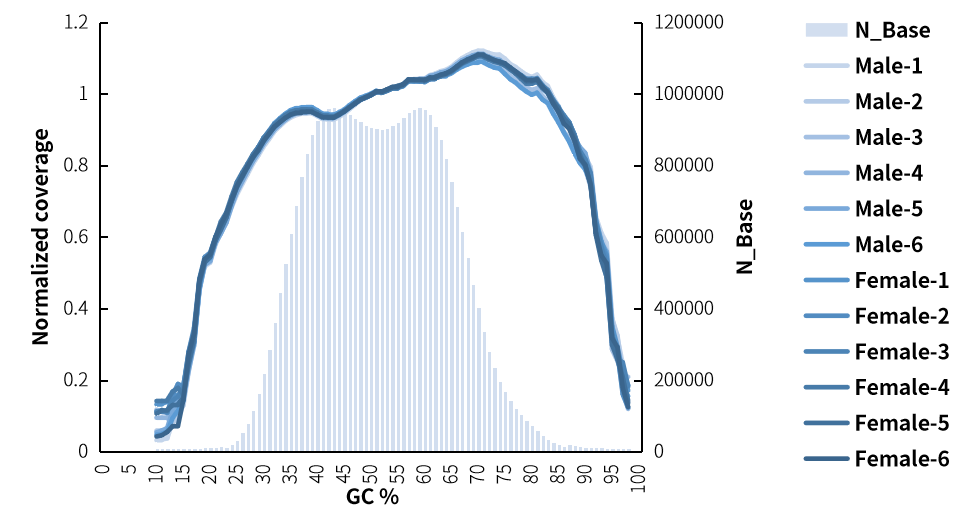
完整标题:NEXome 多杂表现如何?
以下是 12 杂 1 的实际表现(男性 gDNA 和女性 gDNA 各 6 个文库,500 ng/文库,取 25 M reads pair 数据分析)
 |
|
 |
 |
完整标题:NEXome 探针覆盖区域减小了,还可以覆盖外显子侧翼区域吗?
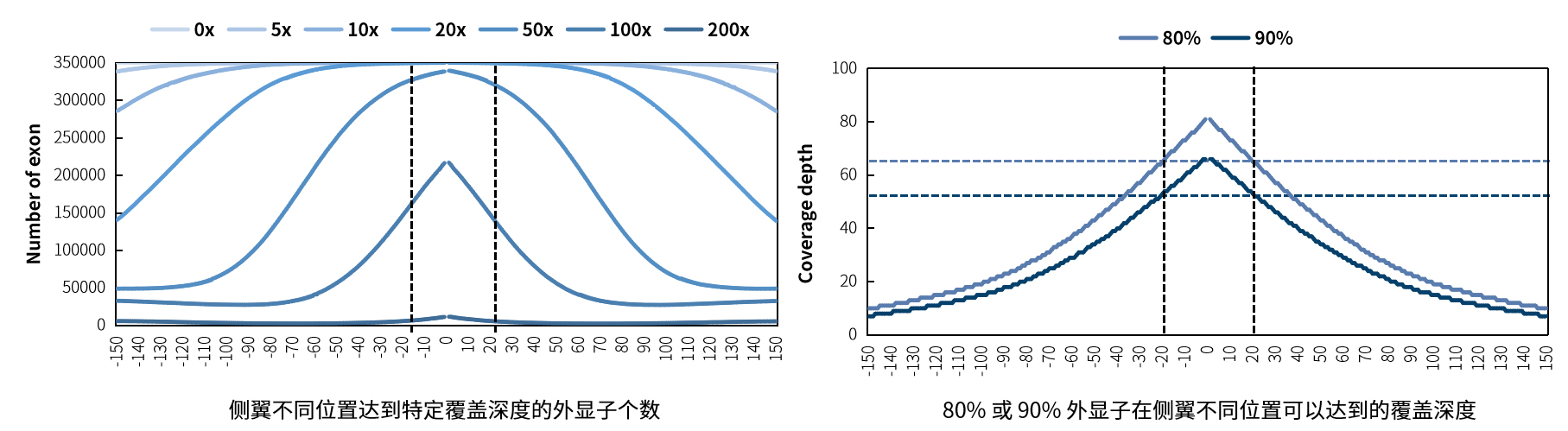
- NEXome 只是减少了不必要的外显子侧翼覆盖,对于侧翼 20 bp 的覆盖是完全可以保证的。
- 如下图所示,测序数据共 10 G,insert size 236 bp 的数据中,99.7% 的外显子侧翼 20 bp 处达到 20x 覆盖,90% 外显子达到 53x,80% 达到 65x。

完整标题:OncoFu Elite (for RNA) Panel v1.0 是否可用于捕获 DNA?
不可以,此 Panel 是基于转录本 cDNA 序列进行探针设计,不兼容 DNACap。


