产品详情

NanOnco Plus Panel v3.0
#1001111F (96 rxn)
- 产品详情
- 产品组分
- 常见问题
- 订购信息
- 资料下载
产品表现
捕获表现
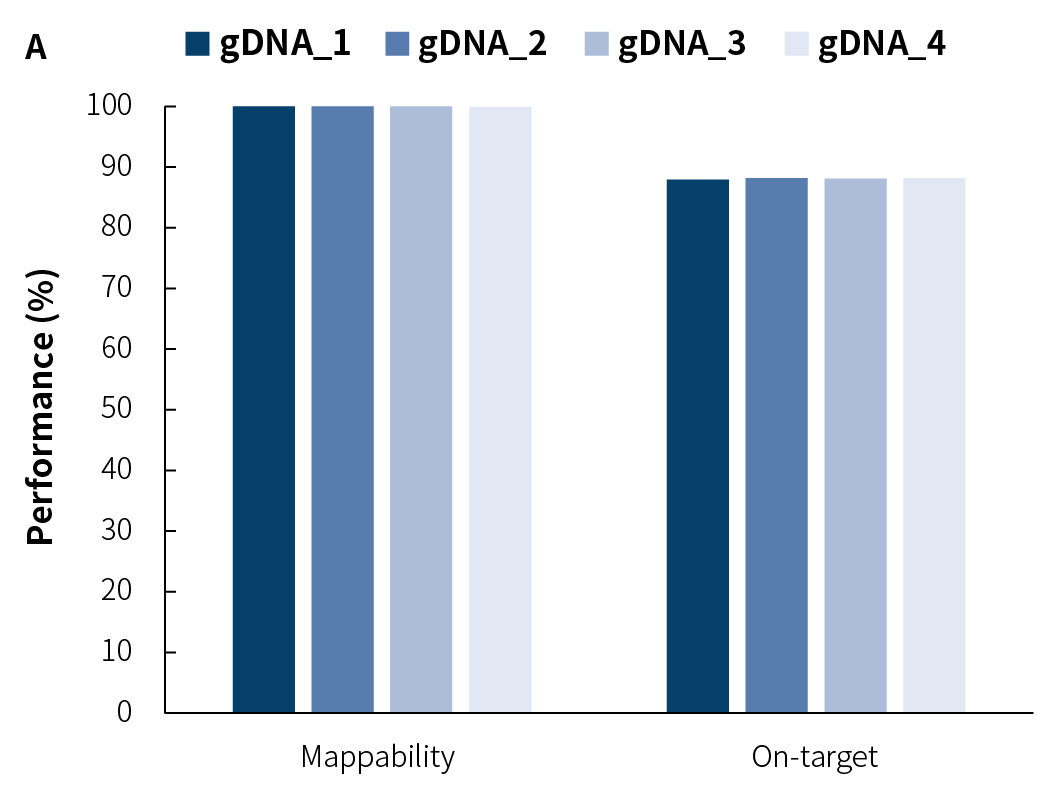
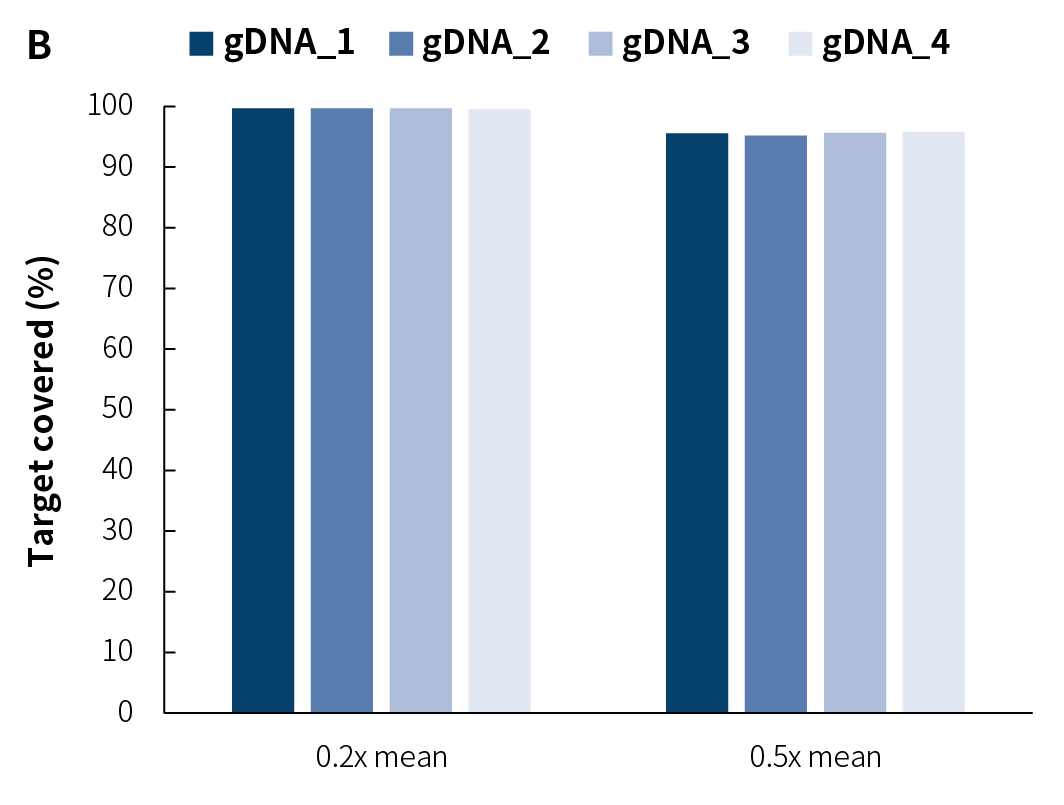
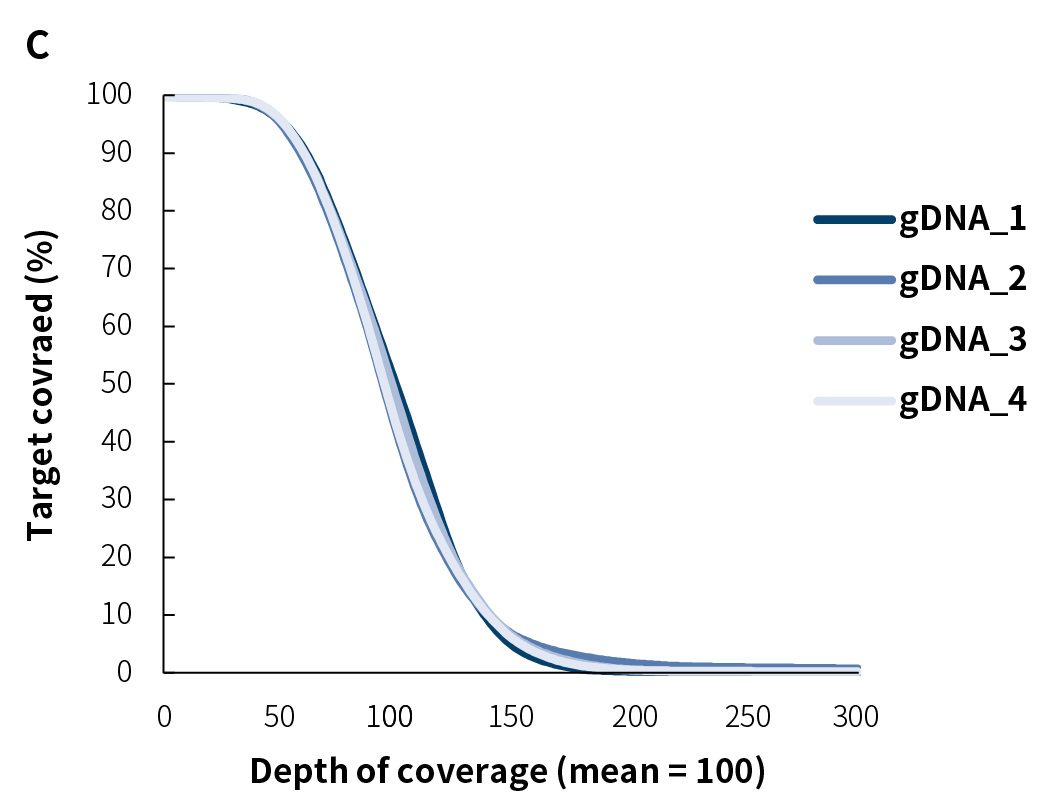
图 1. NanOnco Plus Panel v3.0 对不同 gDNA 文库的捕获表现。利用 NadPrep®快速 DNA 酶切文库构建试剂盒搭配 NadPrep® UDI Adapter (for Illumina®) 构建文库,测序模式为 Illumina Novaseq 6000,PE150,利用 BWA 比对到参考基因组 hg38,按照 reads 数计算。A. 捕获数据比对率和捕获效率;B. 靶区域覆盖度;C. 覆盖均一性和一致性。
注:样本 gDNA_1-4 分别为:人类男性基因组 DNA (Promega-male, G1471);肿瘤结构变异 5% gDNA 标准品 (菁良,GW-OGTM001);肿瘤 SNV 1-25% gDNA 标准品(菁良,GW-OGTM004);肿瘤 SNV 野生型 gDNA 标准品 (菁良,GW-OGTM005)。
多种变异分析
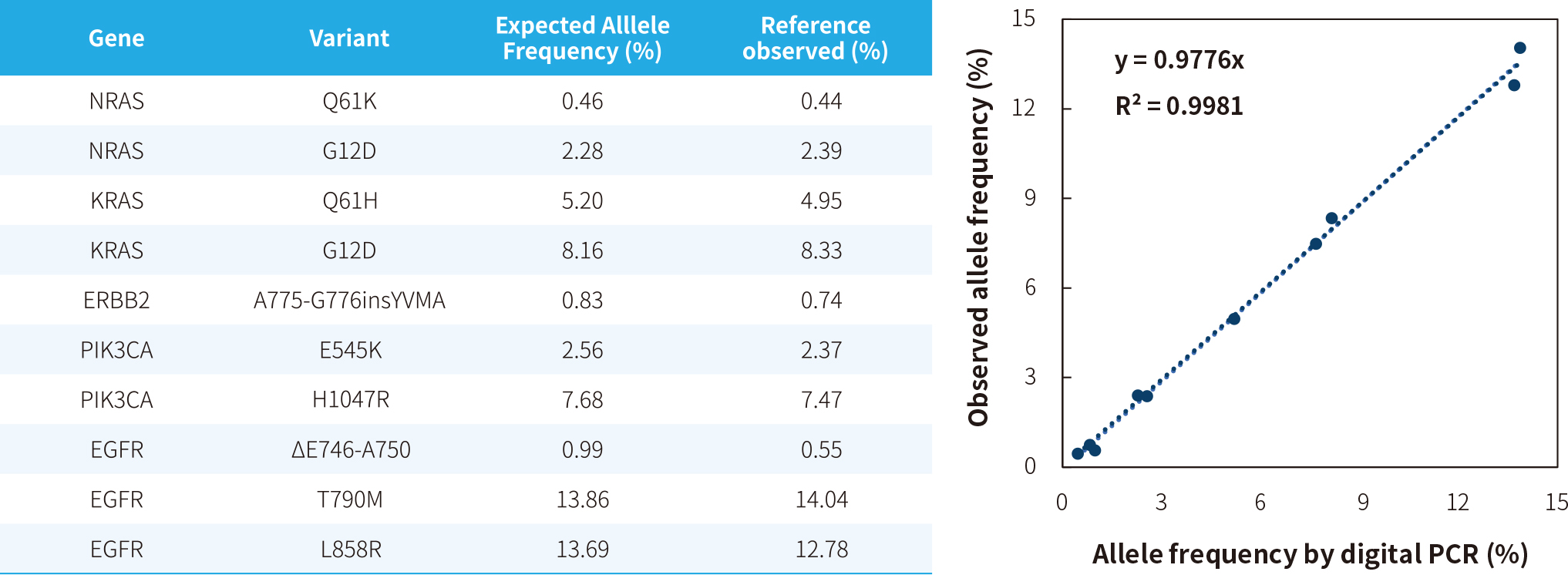
图2. NanOnco Plus Panel v3.0 捕获数据中变异频率与标准品标称频率的一致性展示。利用 NadPrep® 快速 DNA 酶切文库构建试剂盒搭配 NadPrep® Universal Stubby Adapter (UDI) 构建文库,测序模式为 Illumina Novaseq 6000,PE150, Vardict 进行变异分析。
注:样本为 Onco Multiplex gDNA (LDTBIO,定制标准品),平均测序深度 3518x。
表 1. NanOnco Plus Panel v3.0 对其他变异类型检测。
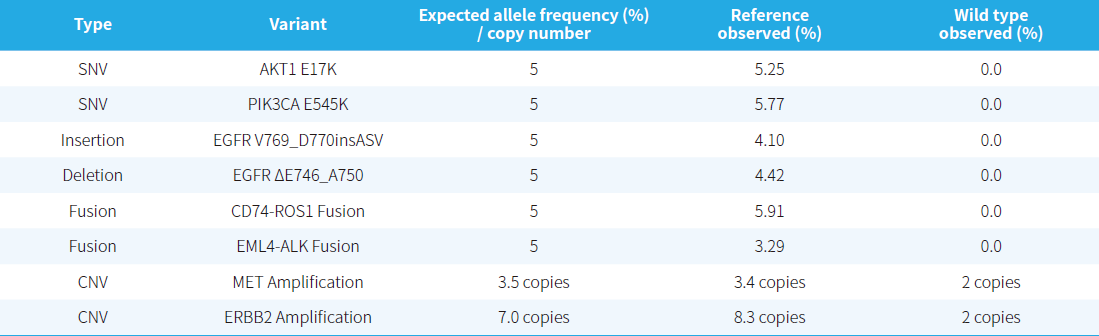
利用 NadPrep® 快速 DNA 酶切文库构建试剂盒搭配 NadPrep® UDI Adapter (for Illumina®) 构建文库,测序模式为 Illumina Novaseq 6000,PE150, Vardict、Delly、CNVkit 分别进行变异分析。GW-OGTM001 的平均测序深度 966.71x,GW-OGTM005 的平均测序深度 1126.04x。
注:样本为肿瘤结构变异 5% gDNA 标准品 (菁良,GW-OGTM001) ,对照为肿瘤 SNV 野生型 gDNA 标准品 (菁良,GW-OGTM005)。
|
货号 |
管盖 颜色 |
产品名称 |
包装 体积 |
包装/储存温度 |
|
1001112F |
|
NanOnco Plus Panel v3.0, 16 rxn |
70 μL |
–20℃ |
|
1001111F |
|
NanOnco Plus Panel v3.0, 96 rxn |
415 μL |
–20℃ |
转录组测序 (RNA-Seq) 文库制备一般分为常规性和链特异性两类,其核心差异在于是否在测序文库中保留转录本的方向性信息。其中,链特异性文库在制备过程中保留了转录本的方向性,测序与下游分析时可判定转录本来源于 DNA 的正义链还是反义链;而常规性文库在 cDNA 合成过程中会丢失方向性信息,测序数据无法直接区分转录方向。与常规性 RNA-Seq 相比,链特异性 RNA-Seq 更能准确地统计转录本数量,明确基因结构,同时识别反义转录本以及发现更多新转录本,因此是研究基因结构与表达调控的重要手段。总体而言,文库制备方法的选择应基于实验目的、成本预算及参考转录组的可用性等因素;若需获取转录本方向性信息,应优先选择链特异性文库。无论选择何种方案,均应严格按照试剂盒使用说明书执行相应操作以确保数据质量。
|
产品 |
货号 |
|
NanOnco Plus Panel v3.0, 16 rxn |
1001112F |
|
NanOnco Plus Panel v3.0, 96 rxn |
1001111F |
产品资料
-
[产品目录] -
产品目录
点击下载
-
[使用说明] -
DNA 文库杂交捕获(Illumina 平台)操作指南 v2.7
点击下载
-
[使用说明] -
DNA 文库杂交捕获 (MGI 平台) 操作指南 v1.8
点击下载
-
[彩页] -
加强型泛癌 panel | NanOnco Plus Panel v3.0
点击下载
-
[Bed文件] -
NanOnco Plus Panel v3.0
点击下载
-
[使用说明] -
NanOnco Plus Panel v3.0 随货说明v1.1
点击下载
-
[MSDS] -
NanOnco Plus Panel v3.0, 96 rxn
点击下载
-
[MSDS] -
NanOnco Plus Panel v3.0, 16 rxn
点击下载
应用指南
DEMO数据
我们的工作人员将在1个工作日内与您取得联系。
如果有任何问题,欢迎联系
400 8717 699 或 support@njnad.com。








